研究建立20L实验室填埋反应器,填埋垃圾参照本地填埋场垃圾组成,并运行220余天,其中整合子基因(intl1)与sulR均呈现显著上升趋势,而blaR类ARGs则有显著下降,从5.0 log10(ARGs/ngDNA) 逐渐降为2.0 log10(ARGs/ngDNA)。通过与环境因子拟合发现,渗滤液中的sulR的丰度主要受到pH值和氮类化合物含量的影响,sulR abundance=log10(intl1 abundance)*0.15 + TN*0.00091- NH3-N*0.0014 + 2.6+ pH*0.19,且该拟合结果与现场显著一致,比简单的sulR-intl拟合程度更高(图2)。而blaR类基因则主要受到环境中COD的影响较大,通过LOESS模拟可以实现准确预测,且预测结果与现场显著一致(P = 0.57)。
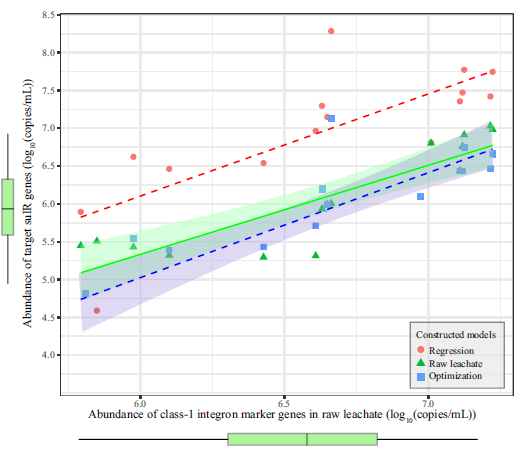
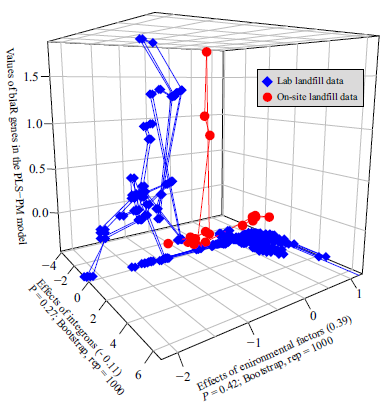
将COD与其他影响因子带入实际渗滤液进行分析,可发现该拟合方式和因子的影响不变,说明该模型可用于实际填埋环境ARGs的预测评估(图-3)。该研究结果,是首次针对渗滤液ARGs的丰度精准预测的尝试,且该方法运用的预测因子,均为常规碳氮检测指标,该结果对环境ARGs预测评估模型进一步发展的指导意义,并对ARGs的风险定量评估有了积极推动作用。
全文链接地址:https://doi.org/10.1016/j.jenvman.2019.04.026
该论文于2019年7月发表在SCI 2 区刊物Journal of Environmental Management (影响因子:4.00)上。
我学院武冬为第一作者,谢冰为通讯作者。